近日台灣疫情再度升溫,儘管疫苗已經開始施打,但隨著COVID-19病毒持續變異,傳播能力也不斷上升,未來對於病毒的研究更顯重要。科學家從語言模型的角度出發,解析如何將原本用於學習人類語言的 AI 語言模型,應用來學習病毒變異的語言,或許將有助未來病毒疫苗的設計。
今年初,MIT 的研究團隊在科學雜誌上發表了一篇有趣的文章,宣稱透過學習人類語言的 AI 語言模型,能夠預測病毒逃逸的潛力。無論是從技術工程的角度或是商業經營的面向分析,都有值得讀者探討的地方。資料科學家將從語言模型的角度出發,帶領讀者一探病毒與人工智慧帶來的啟發。
病毒是甚麼?
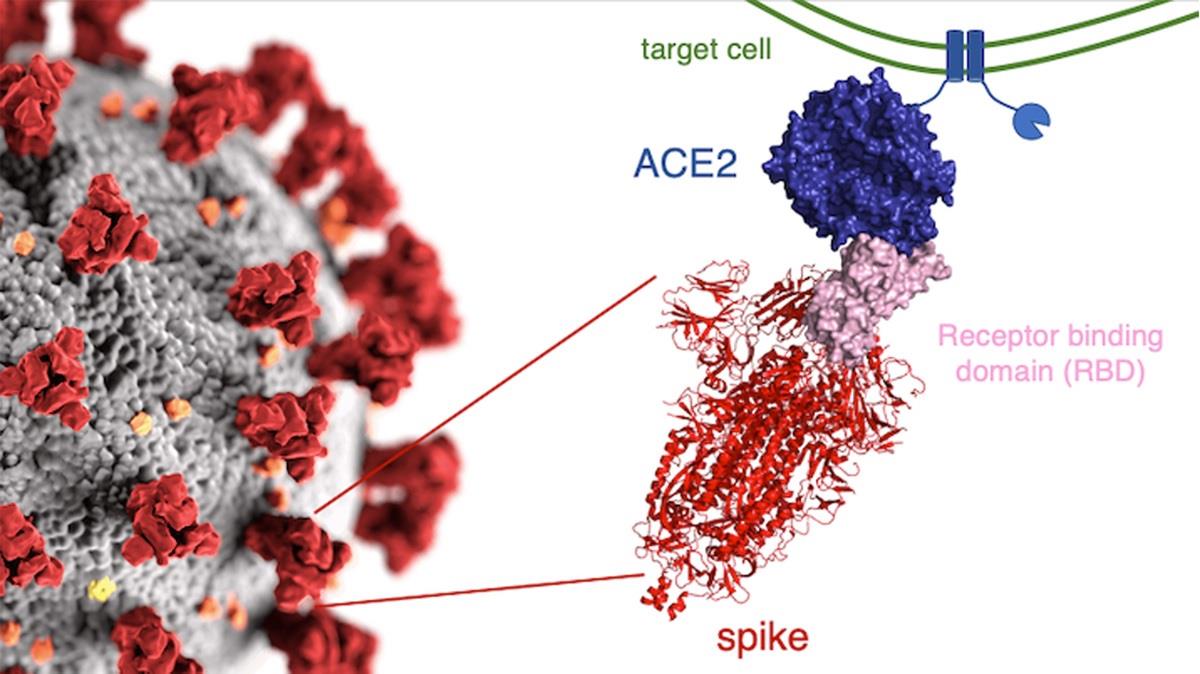
近來新冠病毒 (SARS-CoV-2,如 Fig. 1) 帶著超強的感染力肆虐全球,整個文明社會幾乎全面停擺,可說是聞病毒色變,人們遭受到前所未見的打擊。病毒到底是甚麼呢?簡單來說,它是由一個核酸分子與蛋白質構成的類生物型態,必須藉由感染的機制利用宿主的細胞系統不斷自我複製以延續「自我」的存在。然而,因為不停地複製,產生突變 (mutation) 在所難免:有時是某些胺基酸沒按照藍圖造出來,導致某些重要的蛋白質功能失效,病毒因此掛掉;又或許雖然某個胺基酸弄錯,最終形成的蛋白質依然無礙,病毒持續演化。但有時突變也可能帶來可怕的「幸運」,不僅沒因某個胺基酸弄錯而掛掉,還能逃過人體免疫系統的追捕。當病毒因為基因序列突變導致該病毒抗體或疫苗無法辨識,人們稱之為「病毒逃逸」(viral escape)。
所以當突變不可避免,病毒的生存進化與人體免疫系統的防禦就變成零和遊戲,看誰能笑到最後。顯然人類不會束手就擒,特別是在基因組定序、結構生物學、公衛及流行病防治等領域早已投入大量的努力以累積對抗病毒的本錢。在今年初,來自 MIT 的研究團隊在科學雜誌上發表了一篇很有趣的文章 [2],宣稱能夠預測病毒逃逸的潛力,而方法居然是來自學習人類語言的 AI 語言模型。
奇妙的類比
重大的發現往往來自好奇且具啟發性的問題。「我們能不能將病毒蛋白的基因序列想像成類似人類的自然語言?」MIT 的研究團隊為了回答這個問題,他們做了一個簡單的類比:假設病毒序列就如同人類語言,其上的突變/變異若要存活下去繼續走在演化的道路上就必須符合語言文法的要求或換個說法 fitness 要高;此外,一個病毒「語句」除了要合文法性 (grammaticality) 也要考慮語意 (semantics)。經突變的病毒「語句」即使符合文法,也不代表能夠逃過人體免疫系統的追捕,惟當變異後的病毒「語句」同時大幅改變了語意才能完成病毒逃逸。舉例來說,下面三句話都符合中文文法:
- 種香蕉的農夫說他們這季有好的收穫 (原句)
- 種香蕉的農夫說他們這季有不錯的收穫 (變異)
- 種香蕉的農夫說他們這季有差勁的收穫 (變異)
但是,第 2 句跟原句語意相似,而只有最後一句才大幅改變了語意,具有較高的逃逸潛力。這種搜尋病毒逃逸潛力的方法稱作「受限的語意改變搜索」 [constrained semantic change search (CSCS)],這裡「受限」是指要符合文法。接下來我們將簡單介紹 MIT 研究團隊的文章 [2],了解其病毒的語言模型是如何建立的,而它們又帶給我們甚麼啟發。
病毒可以這樣讀
要建立一個以機器學習為基礎的語言模型,那當然就需要準備大量的文本資料,從大量資料中找出規則。文本裡面的基本單位一般是字詞,通常是字串 (string),也就是一串字元 (character)型式,不能直接用來訓練機器學習模型。因此,為了轉成數值輸入一種簡單的方法是針對常用字詞建立字典,使每個字詞對應一個 token (整數),然後將每個 token 用維度大小為字典長度的 one-hot encoded vector 來表示。例如一個只有三個字詞的字典「我」、 「是」、 「貓奴」,假設 token 分別是 0, 1, 2,那麼 0 (”我”) 的one-hot encoded vector=(1, 0, 0),token 1 是 (0, 1,0),token 2 則為 (0, 0, 1)。然而這種轉換方式當字典龐大時,除了有維度魔咒的問題外,更是毫無字詞間相互關係的意義在裡面。所以近年來自然語言處理最有效率的做法就是利用神經網路來訓練詞嵌入 (word embedding),亦即將每個字詞(或 token) 轉換成一個遠小於字典大小的固定維度向量表徵 (vector representation),不同 vector 間的空間關係會對應到字與其他字間的相對關係,從某個角度來講就是賦予了字義 [3]。把它們投射到三維空間中,可能呈現這樣的關係圖 (Fig. 2):
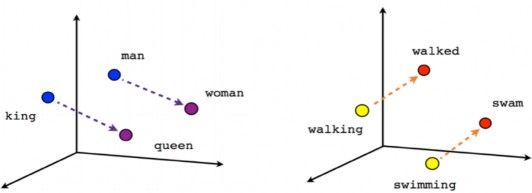
從Fig. 2 可看到king 跟queen 的關係(空間距離)就好比man 跟woman 的關係;同樣的道理,現在進行式walking 跟過去式walked 的關係(空間距離)就好比swimming 跟swam 的關係。
那麼回到病毒「語言」,我們該以甚麼當作文本資料呢?MIT 的研究團隊將焦點聚集在這三種病毒蛋白:新冠病毒棘蛋白 [SARS-CoV-2 spike glycoprotein (Spike)] 、 A 型流感醣蛋白 [influenza A hemagglutinin (HA)] 以及艾滋病毒醣蛋白 [HIV-1 envelope glycoprotein (Env)]。它們都位於病毒表面扮演著感染宿主細胞的關鍵角色,不僅是抗體的鎖定目標也常常是藥物或疫苗的目標。所以,很自然地建造這些病毒蛋白的藍圖便可以當作機器學習需要的文本資料。以目前病毒蛋白質資料庫來說,除了有胺基酸構成的蛋白質序列外,通常還包含了蛋白質摺疊後的三維結構資料。那選哪種訊息當作病毒「語言」最小單位呢?文章的作者認為因為進化的語言主要反映在序列上的變異,所以更準確來說,針對各個病毒來自不同物種的蛋白質序列將是我們的文本資料;其中最小單位就是胺基酸分子。舉例來說,在Influenza Research Database 資料庫中 [4],若將流感 HA 其中數筆訓練資料 (序列中胺基酸以字母編碼 [5])印出來,看起來就像由許多「字母」串成列 (Fig. 3):

再將序列利用胺基酸字典轉為整數token 後,我們同樣可以利用詞嵌入的概念作為訓練資料的輸入。因此接下來我們需要建立語言模型,利用機器學習演算法來幫助我們了解病毒的語言。
深度學習建模
該研究建模的目標是:給定一蛋白質序列 X 長度 N 與所關心變異的位置 i,訓練好的病毒語言模型能夠預測該位置填入某個胺基酸的機率,寫成 p (xi | X[N] \ {i});同時也希望能從中間層得出整個序列的語意嵌入 (semantic embedding) 表徵向量,寫成 z (粗體代表向量)。要達成這個目標,作者運用深度學習針對每一種病毒各自建立一個以兩層雙向 (bidirectional) Long-Short Term Memory (LSTM) 為主體的神經網路模型,如 Fig. 4 所示。
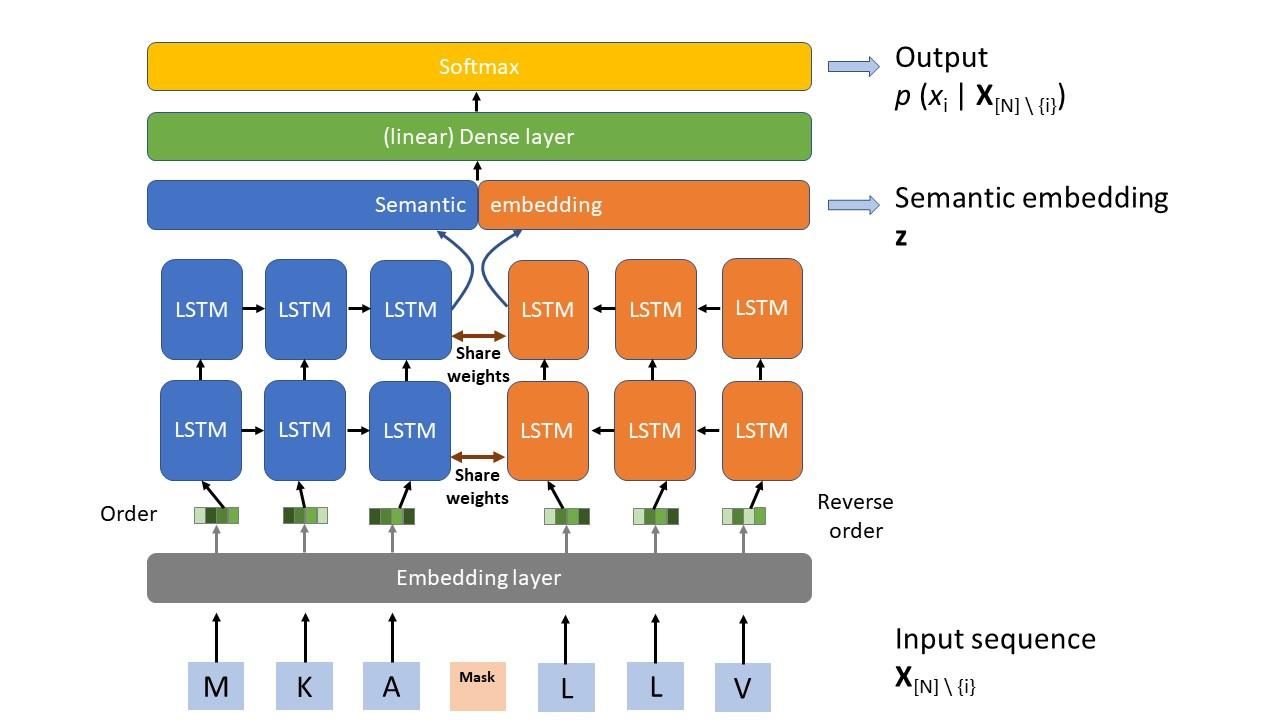
模型並不複雜,蛋白質序列作為輸入後經過 embedding 層轉為適當的向量餵入連續兩層bidirectional LSTM,然後將兩個方向的輸出合併後 (語意嵌入層),再經過一個 dense 層作線性轉換;最後輸出利用 softmax 層得到可能胺基酸的機率分布。LSTM 本身就是神經網路中針對時間序列資料 (在意前後順序) 的常用模組 [6],而對於語言的理解,比如說我們在閱讀一句話的時候,通常不會是逐字分開了解,而是從是句子的前後文來理解句意,那麼使用 LSTM 就不令人意外了。
由於對於病毒的「語言」,無論是語法或是語意都沒有直接的規則或者說正確答案,所以機器學習中常用的監督式學習顯然不是一個好的作法,那麼該如何是好?此時近年來自然語言處理的標準預訓練方法也就是所謂的自監督學習 (self-supervised learning) 便可派上用場 [7]。其概念很簡單,如Fig. 5 所示,對語言文法沒有標準答案沒關係,但羊毛出在羊身上,我們可以利用原來的句子本身。比如說我們可將句子中的某些字遮蔽 (mask),然後訓練模型預測出被遮蔽的字詞!
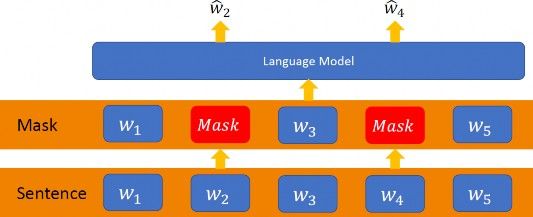
同樣的道理,利用自監督學習,我們可以將輸入的蛋白質序列的任意位置 i 遮蔽,並要求模型預測位置 i 對應的可能胺基酸。當然這時遮蔽的位置肯定是有答案的,所以可以用平時處理分類問題的 categorical cross entropy 來當訓練時的損失函數。這邊有個訓練的細節值得提出來說明一下:我們知道模型的主體是雙向LSTM (Fig. 4),那「雙向」指的是甚麼?從自監督學習中,每個輸入的序列會有一個胺基酸被遮蔽,所以很自然地序列會分成前半段與後半段;此時「雙向」指的就是前半段資料以正序 (頭到尾) 丟入LSTM,而前半段資料以逆序 (尾到頭) 丟入LSTM,然後再將各自輸出合併。這是文章作者試出的最佳訓練法。
訓練好模型後,每個輸入原始序列的語意嵌入表徵向量z,可從語意嵌入層取出。但是因為輸入只接受經遮蔽的序列,所以整個序列的表徵可由從頭到尾輪流遮蔽的 N 個序列分別輸入,最後取平均來代表。當拿來跟某個位置 j 變異的序列的表徵向量算 L1 距離得到 Δz 就可以用來評估 semantic change;同時,將 j 位置變異的序列遮蔽變異位置 j 當輸入,模型的輸出就可得到 grammaticality,p (x’j | X[N] \ {j}),其中 x’代表變異後的胺基酸。原則上將 semantic change與grammaticality 以選定的比例相加就得到該變異CSCS 的最終得分,越高分越有逃逸潛力。
語言模型行不行?
用神經網路模型訓練完後,病毒語言模型到底學到了甚麼,能否得到有意義的推論呢?為此,作者做了數個驗證:
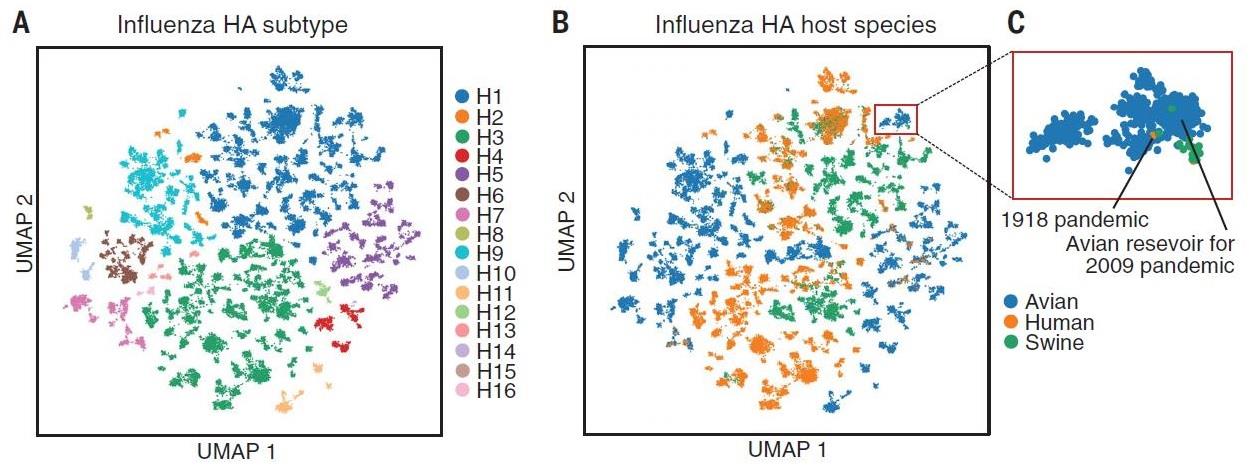
1. 語意嵌入 (semantic embedding) 視覺化
以流感 HA 為例,作者利用非線性降維 Uniform Manifold Approximation and Projection (UMAP) 方法 [8] 將語意嵌入表徵向量 z 投影到二維 (Fig. 6),我們可以很清楚地看到表徵向量能夠適當的區分出不同的子類別或宿主, 甚至也能找到抗原相近但宿主不同的序列(interspecies transmissibility)。
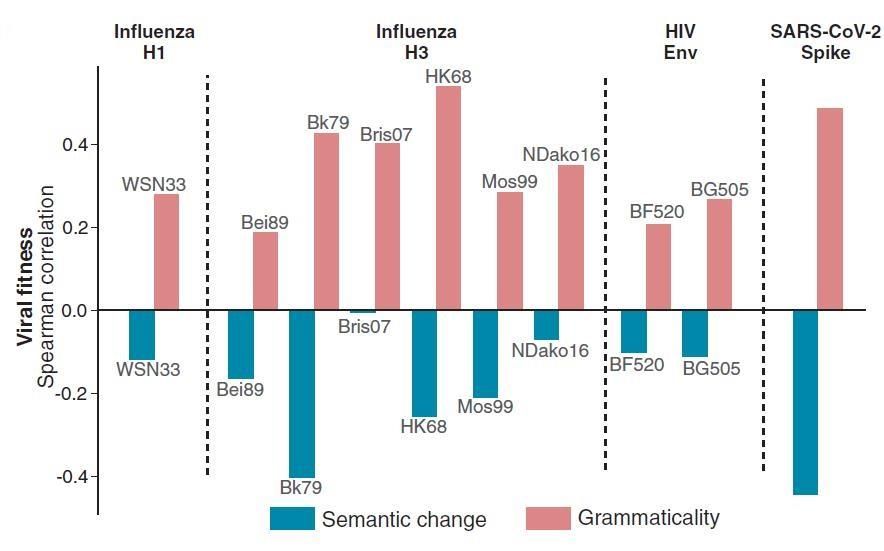
2. 病毒演化的fitness 與語言模型grammaticality/semantic change 相關性
作者針對給定的病毒蛋白用另一個獨立實驗方法 deep-mutational scanning (DMS)考慮數百到數千的變異對蛋白的影響來找出變異的 fitness 分數。從 Spearman correlation 分析中,模型合文法性預測與DMS fitness 顯然成正相關,但語意變化卻呈現弱負相關 (Fig. 7)。後者說明了,當語意變化過大時(違反文法),這種突變可能會導致病毒進化終結,也就是掛掉。不過這也暗示了符合文法性只是蛋白質序列的變異要逃過免疫系統追捕的必要條件,但不充分,或許還需考量語意變化。
3. 病毒逃逸預測與實驗驗證
要驗證達到病毒逃逸的條件須同時滿足合文法性與較大的語意變化,作者用已被實驗證實可造成逃逸的突變結合 DMS 方法來確認哪些是成功逃逸的突變蛋白質序列來對照模型的預測結果。明顯如Fig. 8 所示,同時具有高語意變化與高合文法性確實具較大的病毒逃逸潛力。
4. 逃逸潛力與結構位置的關係
模型純粹從蛋白質序列來學習突變可能造成病毒逃逸的潛力,那麼能不能學到結構相關的模式呢?若以Influenza HA 為例,模型預測告訴我們,逃逸的高潛力發生在頭端而非莖端,正好與實驗結果相符。另外,以 SARS-CoV-2 Spike 為例,N-terminal domain 與 RBD 部分是具高逃逸潛力的,而 S2 部分則很穩定 (Fig. 9,黃色代表逃逸潛力高)。或許這正暗示著開發疫苗時鎖定 S2 可能是更有持續效力的好目標。
對未來的啟發
世界正值推出COVID-19 疫苗之際,病毒脫逃的研究更顯得十分重要。機器學習自然語言模型的好處是,它可以直接從大量的培訓集中學習語言規則;同樣地,若能在生物環境中使用這種模型來學習病毒變異的進化語言,或許人類就不會一直處於被動挨打的地位。就如同 MIT 電腦科學家Bonnie Berger 說:「這是一個告訴你何時展開調查的工具。隨著新病毒株的出現,我們可以標記出哪些病毒株很可能脫逃而值得研究。」[10] AI 工具讓我們走在病毒變異的前面,甚至可以針對未來的病毒設計疫苗。這麼有趣的跨域技術,是不是讓你/妳心動了呢?
References
[2] Hie et al., Science 371, 284–288 (2021).
[3] Word embedding 的概念可參閱https://wiki.pathmind.com/word2vec.
[4] 流感病毒資料下載處https://www.fludb.org/brc/home.spg?decorator=influenza.
[5] 胺基酸編碼可參閱https://en.wikipedia.org/wiki/Amino_acid.
[6] 詳細說明可參考李宏毅老師的課程https://www.youtube.com/watch?v=xCGidAeyS4M.
[7] Jacob Devlin et al., “BERT: Pre-training of Deep Bidirectional Transformers for Language Understanding”, arXiv:1810.04805 (2018).
[8] L. McInnes and J. Healy, “UMAP: Uniform Manifold Approximation and Projection for dimension reduction”, arXiv:1802.03426 (2018).
[9]Brian Hie et al., “ Learning the language of viral evolution and escape”, bioRxiv doi : 10.1101/2020.07.08.193946 (2020).
[10]https://spectrum.ieee.org/the-human-os/biomedical/devices/ai-predicts-most-potent-covid-19-mutations
快速掌握病毒變異和人類語言之間的關係
知道了「病毒的語言模型如何建立的?」之後,你是否也好奇,為何病毒的變異跟人類的語言學有關係呢?或是還不了解模型的人,也可以先看看語言學家這一篇:《白話解讀:為什麼AI應用語言學觀點可以提早捉到病毒變異?》